Три способа хранения и доступа к большому количеству изображений в Python
Оглавление
- Установка
- Хранение одного изображения
- Хранение многих изображений
- Чтение одного изображения
- Чтение многих изображений
- Рассмотрение использования диска
- Дискуссия
Почему вы хотите узнать больше о различных способах хранения и доступа к изображениям в Python? Если вы сегментируете горстку изображений по цвету или определяете лица по одному с помощью OpenCV, то вам не нужно об этом беспокоиться. Даже если вы используете Python Imaging Library (PIL) для рисования на нескольких сотнях фотографий, вам это все равно не нужно. Хранение изображений на диске, в виде .png или .jpg файлов, вполне уместно и целесообразно.
Однако количество изображений, необходимых для решения той или иной задачи, становится все больше и больше. Такие алгоритмы, как конволюционные нейронные сети, также известные как конвнеты или CNN, могут обрабатывать огромные наборы изображений и даже обучаться на них. Если вам интересно, вы можете прочитать о том, как конвентные сети могут быть использованы для ранжирования селфи или для анализа настроений.
ImageNet - известная общедоступная база данных изображений, созданная для обучения моделей в таких задачах, как классификация, обнаружение и сегментация объектов, и включающая более 14 миллионов изображений.
Подумайте о том, сколько времени потребуется, чтобы загрузить их все в память для обучения, причем партиями, возможно, сотни или тысячи раз. Продолжайте читать, и вы убедитесь, что это займет довольно много времени - по крайней мере, достаточно, чтобы отойти от компьютера и заняться другими делами, пока вы мечтаете работать в Google или NVIDIA.
В этом уроке вы узнаете о:
- Хранение изображений на диске в виде
.pngфайлов - Хранение изображений в базах данных с молниеносным отображением памяти (LMDB)
- Хранение изображений в иерархическом формате данных (HDF5)
Вы также изучите следующее:
- Почему стоит рассмотреть альтернативные методы хранения данных
- Каковы различия в производительности при чтении и записи отдельных изображений
- Каковы различия в производительности при чтении и записи многих изображений
- Как три метода сравниваются по использованию диска
Если ни один из методов хранения данных вам ничего не напоминает, не волнуйтесь: для этой статьи вам понадобится лишь достаточно прочная основа Python и базовое понимание изображений (что они действительно состоят из многомерных массивов чисел) и относительной памяти, например, разницы между 10 МБ и 10 ГБ.
Давайте начнем!
Настройка
Вам понадобится набор данных изображений для экспериментов, а также несколько пакетов Python.
Набор данных для игры
Мы будем использовать набор данных изображений Канадского института передовых исследований, более известный как CIFAR-10, который состоит из 60 000 цветных изображений размером 32x32 пикселя, относящихся к различным классам объектов, таким как собаки, кошки и самолеты. Относительно CIFAR - не очень большой набор данных, но если бы мы использовали полный набор данных TinyImages, то вам потребовалось бы около 400 ГБ свободного дискового пространства, что, вероятно, стало бы ограничивающим фактором.
За создание набора данных, описанного в главе 3 этого технического отчета, спасибо Алексу Крижевскому, Виноду Наиру и Джеффри Хинтону.
Если вы хотите следовать примерам кода в этой статье, вы можете скачать CIFAR-10 здесь, выбрав версию Python. Вы пожертвуете 163 МБ дискового пространства:
Изображение: А. Крижевский
Когда вы скачаете и распакуете папку, вы обнаружите, что файлы не являются человекочитаемыми файлами изображений. На самом деле они были сериализованы и сохранены партиями с помощью cPickle.
Хотя мы не будем рассматривать pickle или cPickle в этой статье, кроме как для извлечения набора данных CIFAR, стоит упомянуть, что модуль Python pickle имеет ключевое преимущество - возможность сериализовать любой объект Python без дополнительного кода или преобразований с вашей стороны. У него также есть потенциально серьезный недостаток, заключающийся в том, что он представляет риск для безопасности и плохо справляется с очень большими объемами данных.
Следующий код распаковывает каждый из пяти пакетных файлов и загружает все изображения в массив NumPy:
import numpy as np
import pickle
from pathlib import Path
# Path to the unzipped CIFAR data
data_dir = Path("data/cifar-10-batches-py/")
# Unpickle function provided by the CIFAR hosts
def unpickle(file):
with open(file, "rb") as fo:
dict = pickle.load(fo, encoding="bytes")
return dict
images, labels = [], []
for batch in data_dir.glob("data_batch_*"):
batch_data = unpickle(batch)
for i, flat_im in enumerate(batch_data[b"data"]):
im_channels = []
# Each image is flattened, with channels in order of R, G, B
for j in range(3):
im_channels.append(
flat_im[j * 1024 : (j + 1) * 1024].reshape((32, 32))
)
# Reconstruct the original image
images.append(np.dstack((im_channels)))
# Save the label
labels.append(batch_data[b"labels"][i])
print("Loaded CIFAR-10 training set:")
print(f" - np.shape(images) {np.shape(images)}")
print(f" - np.shape(labels) {np.shape(labels)}")
Теперь все изображения находятся в оперативной памяти в переменной images , а их соответствующие метаданные - в labels, и они готовы к работе с ними. Далее вы можете установить пакеты Python, которые вы будете использовать для трех методов.
Примечание: В последнем блоке кода использовались f-строки. Подробнее о них вы можете прочитать в Python's F-String for String Interpolation and Formatting.
Настройка хранения изображений на диске
Вам нужно будет настроить свое окружение на стандартный метод сохранения и доступа к этим изображениям с диска. В этой статье предполагается, что в вашей системе установлен Python 3.x, и для работы с изображениями будет использоваться Pillow:
$ pip install Pillow
Альтернативно, если вы предпочитаете, вы можете установить его с помощью Anaconda:
$ conda install -c conda-forge pillow
Примечание: PIL - это оригинальная версия библиотеки Python Imaging Library, которая больше не поддерживается и не совместима с Python 3.x. Если у вас ранее была установлена PIL, обязательно удалите ее перед установкой Pillow, поскольку они не могут существовать вместе.
Теперь вы готовы к хранению и чтению изображений с диска.
Начало работы с LMDB
LMDB, иногда называемая "Lightning Database", означает Lightning Memory-Mapped Database, потому что она быстрая и использует файлы, отображаемые в памяти. Это хранилище ключевых значений, а не реляционная база данных.
С точки зрения реализации, LMDB представляет собой дерево B+, что в основном означает, что это древовидная графовая структура, хранящаяся в памяти, где каждый элемент ключ-значение является узлом, а узлы могут иметь много дочерних элементов. Узлы одного уровня связаны друг с другом для быстрого обхода.
Критически важно, что ключевые компоненты дерева B+ устанавливаются в соответствии с размером страницы операционной системы хоста, что максимизирует эффективность при обращении к любой паре ключ-значение в базе данных. Поскольку высокая производительность LMDB в значительной степени зависит от этого конкретного момента, было показано, что эффективность LMDB зависит от базовой файловой системы и ее реализации.
Еще одной ключевой причиной эффективности LMDB является то, что она отображается на память. Это означает, что она возвращает прямые указатели на адреса памяти как ключей, так и значений, без необходимости копировать что-либо в память, как это делают большинство других баз данных.
Те, кто хочет немного больше узнать о внутренних деталях реализации B+ деревьев, могут ознакомиться с этой статьей о B+ деревьях, а затем поиграть с этой визуализацией вставки узлов.
Если деревья B+ вас не интересуют, не беспокойтесь. Для использования LMDB вам не нужно много знать об их внутренней реализации. Мы будем использовать привязку Python для библиотеки LMDB C, которую можно установить через pip:
$ pip install lmdb
У вас также есть возможность установки через Anaconda:
$ conda install -c conda-forge python-lmdb
Убедитесь, что вы можете import lmdb работать с оболочкой Python, и все готово.
Начало работы с HDF5
HDF5 означает Hierarchical Data Format, формат файлов, упоминаемый как HDF4 или HDF5. Нам не нужно беспокоиться о HDF4, так как HDF5 - это текущая поддерживаемая версия.
Интересно, что HDF зародился в Национальном центре суперкомпьютерных приложений как портативный, компактный формат научных данных. Если вам интересно, широко ли он используется, ознакомьтесь с информацией о HDF5 от НАСА из их проекта Earth Data.
HDF-файлы состоят из двух типов объектов:
- Наборы данных
- Группы
Наборы данных - это многомерные массивы, а группы состоят из наборов данных или других групп. Многомерные массивы любого размера и типа могут храниться в виде набора данных, но размеры и тип должны быть однородными в пределах набора данных. Каждый набор данных должен содержать однородный N-мерный массив. При этом, поскольку группы и наборы данных могут быть вложенными, вы все равно можете получить необходимую вам неоднородность:
$ pip install h5py
Как и в случае с другими библиотеками, вы можете альтернативно установить через Anaconda:
$ conda install -c conda-forge h5py
Если вы можете import h5py из оболочки Python, значит, все настроено правильно.
Хранение одного изображения
Теперь, когда вы получили общее представление о методах, давайте сразу же погрузимся в работу и рассмотрим количественное сравнение основных задач, которые нас интересуют: сколько времени требуется на чтение и запись файлов и сколько дисковой памяти будет использовано. Это также послужит базовым введением в то, как работают методы, с примерами кода, как их использовать.
Когда я говорю о "файлах", я обычно имею в виду большое их количество. Однако важно делать различие, поскольку некоторые методы могут быть оптимизированы для разных операций и количества файлов.
В целях эксперимента мы можем сравнить производительность между различными количествами файлов, в 10 раз от одного изображения до 100 000 изображений. Поскольку наши пять партий CIFAR-10 составляют 50 000 изображений, мы можем использовать каждое изображение дважды, чтобы получить 100 000 изображений.
Для подготовки к экспериментам необходимо создать для каждого метода папку, содержащую все файлы базы данных или изображения, и сохранить пути к этим папкам в переменных:
from pathlib import Path
disk_dir = Path("data/disk/")
lmdb_dir = Path("data/lmdb/")
hdf5_dir = Path("data/hdf5/")
Path не будет автоматически создавать папки для вас, если вы специально не попросите его об этом:
disk_dir.mkdir(parents=True, exist_ok=True)
lmdb_dir.mkdir(parents=True, exist_ok=True)
hdf5_dir.mkdir(parents=True, exist_ok=True)
Теперь можно перейти к выполнению реальных экспериментов с примерами кода, показывающими, как выполнять основные задачи с помощью трех различных методов. Мы можем использовать модуль timeit, который входит в стандартную библиотеку Python, чтобы помочь определить время проведения экспериментов.
Хотя основной целью этой статьи не является изучение API различных пакетов Python, полезно иметь представление о том, как они могут быть реализованы. Мы рассмотрим общие принципы, а также весь код, используемый для проведения экспериментов по хранению данных.
Сохранение на диск
Нашим входом для этого эксперимента является одно изображение image, которое в настоящее время находится в памяти в виде массива NumPy. Вы хотите сначала сохранить его на диске в виде образа .png и назвать его с помощью уникального идентификатора образа image_id. Это можно сделать с помощью пакета Pillow, который вы установили ранее:
from PIL import Image
import csv
def store_single_disk(image, image_id, label):
""" Stores a single image as a .png file on disk.
Parameters:
---------------
image image array, (32, 32, 3) to be stored
image_id integer unique ID for image
label image label
"""
Image.fromarray(image).save(disk_dir / f"{image_id}.png")
with open(disk_dir / f"{image_id}.csv", "wt") as csvfile:
writer = csv.writer(
csvfile, delimiter=" ", quotechar="|", quoting=csv.QUOTE_MINIMAL
)
writer.writerow([label])
Это сохраняет изображение. Во всех реалистичных приложениях вы также заботитесь о метаданных, прикрепленных к изображению, которые в нашем примере набора данных являются меткой изображения. При сохранении изображений на диск существует несколько вариантов сохранения метаданных.
Одним из решений является кодирование меток в имя изображения. Преимущество этого способа в том, что он не требует дополнительных файлов.
Однако у этого способа есть и большой недостаток - он заставляет вас работать со всеми файлами каждый раз, когда вы что-то делаете с этикетками. Хранение меток в отдельном файле позволяет вам играть только с ними, без необходимости загружать изображения. Выше я сохранил метки в отдельных файлах .csv для этого эксперимента.
Теперь перейдем к выполнению точно такой же задачи с помощью LMDB.
Сохранение в LMDB
Во-первых, LMDB - это система хранения ключ-значение, где каждая запись сохраняется в виде массива байтов, поэтому в нашем случае ключами будут уникальные идентификаторы каждого изображения, а значением - само изображение. Ожидается, что и ключи, и значения будут строками, поэтому обычно значение сериализуется как строка, а затем отсериализуется при обратном чтении.
Для сериализации можно использовать pickle. Любой объект Python может быть сериализован, так что вы можете включить метаданные изображения в базу данных. Это избавит вас от необходимости прикреплять метаданные обратно к данным изображения, когда мы будем загружать набор данных с диска.
Вы можете создать базовый Python-класс для изображения и его метаданных:
class CIFAR_Image:
def __init__(self, image, label):
# Dimensions of image for reconstruction - not really necessary
# for this dataset, but some datasets may include images of
# varying sizes
self.channels = image.shape[2]
self.size = image.shape[:2]
self.image = image.tobytes()
self.label = label
def get_image(self):
""" Returns the image as a numpy array. """
image = np.frombuffer(self.image, dtype=np.uint8)
return image.reshape(*self.size, self.channels)
Во-вторых, поскольку LMDB отображается в памяти, новые базы данных должны знать, какой объем памяти они будут использовать. В нашем случае это относительно просто, но в других случаях это может стать большой проблемой, о чем вы узнаете в следующем разделе. LMDB называет эту переменную map_size.
Наконец, операции чтения и записи в LMDB выполняются в transactions. Вы можете считать, что они похожи на операции с традиционной базой данных, состоящие из группы операций над базой данных. Это может показаться уже значительно более сложным, чем дисковая версия, но держитесь и продолжайте читать!
После этих трех пунктов давайте рассмотрим код для сохранения одного изображения в LMDB:
import lmdb
import pickle
def store_single_lmdb(image, image_id, label):
""" Stores a single image to a LMDB.
Parameters:
---------------
image image array, (32, 32, 3) to be stored
image_id integer unique ID for image
label image label
"""
map_size = image.nbytes * 10
# Create a new LMDB environment
env = lmdb.open(str(lmdb_dir / f"single_lmdb"), map_size=map_size)
# Start a new write transaction
with env.begin(write=True) as txn:
# All key-value pairs need to be strings
value = CIFAR_Image(image, label)
key = f"{image_id:08}"
txn.put(key.encode("ascii"), pickle.dumps(value))
env.close()
Примечание: Неплохо бы подсчитать точное количество байт, которое будет занимать каждая пара ключ-значение.
При наборе данных, состоящем из изображений разного размера, это будет приближением, но вы можете использовать sys.getsizeof(), чтобы получить разумное приближение. Помните, что sys.getsizeof(CIFAR_Image) вернет только размер определения класса, который равен 1056, а не размер инстанцированного объекта.
Функция также не сможет полностью вычислить вложенные элементы, списки или объекты, содержащие ссылки на другие объекты.
В качестве альтернативы можно использовать pympler, чтобы сэкономить на вычислениях, определив точный размер объекта.
Теперь вы готовы сохранить изображение в LMDB. Наконец, давайте рассмотрим последний метод, HDF5.
Хранение с помощью HDF5
Помните, что файл HDF5 может содержать более одного набора данных. В этом довольно тривиальном случае можно создать два набора данных, один для изображения, а другой для его метаданных:
import h5py
def store_single_hdf5(image, image_id, label):
""" Stores a single image to an HDF5 file.
Parameters:
---------------
image image array, (32, 32, 3) to be stored
image_id integer unique ID for image
label image label
"""
# Create a new HDF5 file
file = h5py.File(hdf5_dir / f"{image_id}.h5", "w")
# Create a dataset in the file
dataset = file.create_dataset(
"image", np.shape(image), h5py.h5t.STD_U8BE, data=image
)
meta_set = file.create_dataset(
"meta", np.shape(label), h5py.h5t.STD_U8BE, data=label
)
file.close()
h5py.h5t.STD_U8BE задает тип данных, которые будут храниться в наборе данных, в данном случае это беззнаковые 8-битные целые числа. Полный список предопределенных типов данных HDF можно посмотреть здесь.
Примечание: Выбор типа данных сильно влияет на время выполнения и требования к хранению данных HDF5, поэтому лучше всего выбирать минимальные требования.
Теперь, когда мы рассмотрели три способа сохранения одного изображения, давайте перейдем к следующему шагу.
Эксперименты по хранению одного изображения
Теперь вы можете поместить все три функции для сохранения одного изображения в словарь, который можно будет вызвать позже во время экспериментов с таймингом:
_store_single_funcs = dict(
disk=store_single_disk, lmdb=store_single_lmdb, hdf5=store_single_hdf5
)
Наконец, все готово для проведения эксперимента с таймером. Давайте попробуем сохранить первое изображение из CIFAR и соответствующую ему метку, а также сохранить его тремя различными способами:
from timeit import timeit
store_single_timings = dict()
for method in ("disk", "lmdb", "hdf5"):
t = timeit(
"_store_single_funcs[method](image, 0, label)",
setup="image=images[0]; label=labels[0]",
number=1,
globals=globals(),
)
store_single_timings[method] = t
print(f"Method: {method}, Time usage: {t}")
Примечание: Во время работы с LMDB вы можете увидеть MapFullError: mdb_txn_commit: MDB_MAP_FULL: Environment mapsize limit reached ошибку. Важно отметить, что LMDB не перезаписывает ранее существовавшие значения, даже если они имеют одинаковый ключ.
Это способствует быстрому времени записи, но также означает, что если вы сохраняете изображение более одного раза в одном и том же LMDB-файле, то вы израсходуете весь размер карты. Если вы запускаете функцию store, не забудьте сначала удалить все существующие LMDB-файлы.
Помните, что нас интересует время выполнения, отображаемое здесь в секундах, а также использование памяти:
| Method | Save Single Image + Meta | Memory |
|---|---|---|
| Disk | 1.915 ms | 8 K |
| LMDB | 1.203 ms | 32 K |
| HDF5 | 8.243 ms | 8 K |
Здесь есть два вывода:
- Все методы тривиально быстрые.
- С точки зрения использования диска, LMDB использует больше.
Очевидно, что, несмотря на небольшое превосходство LMDB в производительности, мы так и не убедили никого в том, почему не стоит хранить изображения на диске. В конце концов, это человекочитаемый формат, и вы можете открывать и просматривать их из любого браузера файловой системы! Что ж, пришло время посмотреть на гораздо большее количество изображений...
Хранение большого количества изображений
Вы видели код для использования различных методов хранения для сохранения одного изображения, теперь нам нужно настроить код для сохранения множества изображений, а затем запустить эксперимент с таймером.
Настройка кода для многих изображений
Сохранение нескольких изображений в виде .png файлов - это простое действие, как вызов store_single_method() несколько раз. Но это не верно для LMDB или HDF5, поскольку вам не нужен отдельный файл базы данных для каждого изображения. Скорее, вы хотите поместить все изображения в один или несколько файлов.
Вам нужно будет немного изменить код и создать три новые функции, принимающие несколько изображений, store_many_disk(), store_many_lmdb() и store_many_hdf5:
store_many_disk(images, labels):
""" Stores an array of images to disk
Parameters:
---------------
images images array, (N, 32, 32, 3) to be stored
labels labels array, (N, 1) to be stored
"""
num_images = len(images)
# Save all the images one by one
for i, image in enumerate(images):
Image.fromarray(image).save(disk_dir / f"{i}.png")
# Save all the labels to the csv file
with open(disk_dir / f"{num_images}.csv", "w") as csvfile:
writer = csv.writer(
csvfile, delimiter=" ", quotechar="|", quoting=csv.QUOTE_MINIMAL
)
for label in labels:
# This typically would be more than just one value per row
writer.writerow([label])
def store_many_lmdb(images, labels):
""" Stores an array of images to LMDB.
Parameters:
---------------
images images array, (N, 32, 32, 3) to be stored
labels labels array, (N, 1) to be stored
"""
num_images = len(images)
map_size = num_images * images[0].nbytes * 10
# Create a new LMDB DB for all the images
env = lmdb.open(str(lmdb_dir / f"{num_images}_lmdb"), map_size=map_size)
# Same as before — but let's write all the images in a single transaction
with env.begin(write=True) as txn:
for i in range(num_images):
# All key-value pairs need to be Strings
value = CIFAR_Image(images[i], labels[i])
key = f"{i:08}"
txn.put(key.encode("ascii"), pickle.dumps(value))
env.close()
def store_many_hdf5(images, labels):
""" Stores an array of images to HDF5.
Parameters:
---------------
images images array, (N, 32, 32, 3) to be stored
labels labels array, (N, 1) to be stored
"""
num_images = len(images)
# Create a new HDF5 file
file = h5py.File(hdf5_dir / f"{num_images}_many.h5", "w")
# Create a dataset in the file
dataset = file.create_dataset(
"images", np.shape(images), h5py.h5t.STD_U8BE, data=images
)
meta_set = file.create_dataset(
"meta", np.shape(labels), h5py.h5t.STD_U8BE, data=labels
)
file.close()
Для того чтобы можно было хранить на диске более одного файла, метод image files был изменен, чтобы перебирать каждое изображение в списке. Для LMDB цикл также необходим, поскольку мы создаем объект CIFAR_Image для каждого изображения и его метаданных.
Наименьшая корректировка наблюдается при использовании метода HDF5. На самом деле, здесь вообще нет никакой корректировки! Файлы HFD5 не имеют ограничений на размер файла, кроме внешних ограничений или размера набора данных, поэтому все изображения были помещены в один набор данных, как и раньше.
Далее вам нужно подготовить набор данных для экспериментов, увеличив его размер.
Подготовка набора данных
Перед тем как запустить эксперименты снова, давайте сначала удвоим размер набора данных, чтобы мы могли тестировать до 100 000 изображений:
cutoffs = [10, 100, 1000, 10000, 100000]
# Let's double our images so that we have 100,000
images = np.concatenate((images, images), axis=0)
labels = np.concatenate((labels, labels), axis=0)
# Make sure you actually have 100,000 images and labels
print(np.shape(images))
print(np.shape(labels))
Теперь, когда изображений достаточно, пришло время для эксперимента.
Эксперимент по хранению большого количества изображений
Как и в случае с чтением множества изображений, вы можете создать словарь, обрабатывающий все функции с помощью store_many_, и запустить эксперименты:
_store_many_funcs = dict(
disk=store_many_disk, lmdb=store_many_lmdb, hdf5=store_many_hdf5
)
from timeit import timeit
store_many_timings = {"disk": [], "lmdb": [], "hdf5": []}
for cutoff in cutoffs:
for method in ("disk", "lmdb", "hdf5"):
t = timeit(
"_store_many_funcs[method](images_, labels_)",
setup="images_=images[:cutoff]; labels_=labels[:cutoff]",
number=1,
globals=globals(),
)
store_many_timings[method].append(t)
# Print out the method, cutoff, and elapsed time
print(f"Method: {method}, Time usage: {t}")
Если вы следите за ходом работы и выполняете код самостоятельно, вам придется немного посидеть в напряжении и подождать, пока 111 110 изображений будут сохранены на ваш диск по три раза в трех разных форматах. Вам также придется попрощаться с примерно 2 Гб дискового пространства.
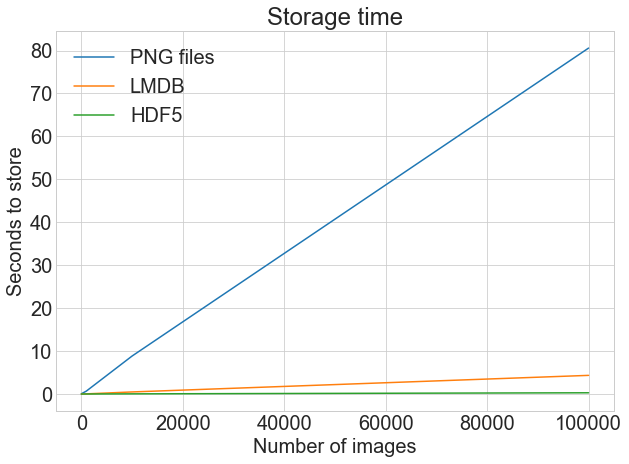
Теперь к моменту истины! Сколько времени заняло все это хранение? Фотография стоит тысячи слов:
На первом графике показано обычное, не скорректированное время хранения, что подчеркивает резкую разницу между хранением в файлах .png и LMDB или HDF5.
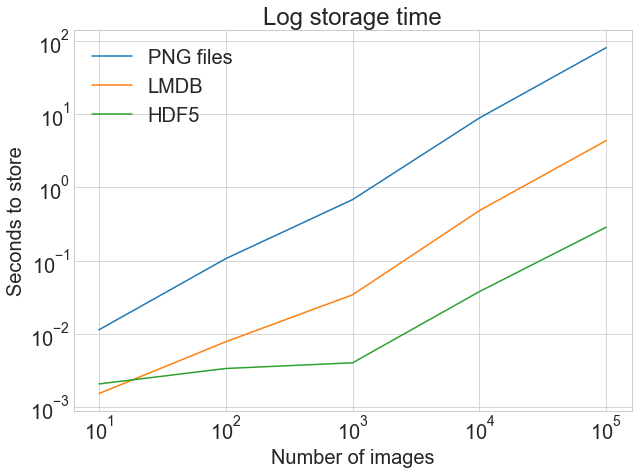
На втором графике показаны log тайминги, подчеркивающие, что HDF5 начинает работать медленнее, чем LMDB, но при увеличении количества изображений выходит немного вперед.
Хотя точные результаты могут отличаться в зависимости от вашей машины, именно поэтому стоит задуматься о LMDB и HDF5. Вот код, сгенерировавший приведенный выше график:
import matplotlib.pyplot as plt
def plot_with_legend(
x_range, y_data, legend_labels, x_label, y_label, title, log=False
):
""" Displays a single plot with multiple datasets and matching legends.
Parameters:
--------------
x_range list of lists containing x data
y_data list of lists containing y values
legend_labels list of string legend labels
x_label x axis label
y_label y axis label
"""
plt.style.use("seaborn-whitegrid")
plt.figure(figsize=(10, 7))
if len(y_data) != len(legend_labels):
raise TypeError(
"Error: number of data sets does not match number of labels."
)
all_plots = []
for data, label in zip(y_data, legend_labels):
if log:
temp, = plt.loglog(x_range, data, label=label)
else:
temp, = plt.plot(x_range, data, label=label)
all_plots.append(temp)
plt.title(title)
plt.xlabel(x_label)
plt.ylabel(y_label)
plt.legend(handles=all_plots)
plt.show()
# Getting the store timings data to display
disk_x = store_many_timings["disk"]
lmdb_x = store_many_timings["lmdb"]
hdf5_x = store_many_timings["hdf5"]
plot_with_legend(
cutoffs,
[disk_x, lmdb_x, hdf5_x],
["PNG files", "LMDB", "HDF5"],
"Number of images",
"Seconds to store",
"Storage time",
log=False,
)
plot_with_legend(
cutoffs,
[disk_x, lmdb_x, hdf5_x],
["PNG files", "LMDB", "HDF5"],
"Number of images",
"Seconds to store",
"Log storage time",
log=True,
)
Теперь перейдем к считыванию изображений обратно.
Чтение одного изображения
Сначала рассмотрим случай чтения одного изображения обратно в массив для каждого из трех методов.
Чтение с диска
Из всех трех методов LMDB требует больше всего работы при чтении файлов изображений из памяти, так как в этом случае требуется сериализация. Давайте рассмотрим эти функции, которые считывают одно изображение для каждого из трех форматов хранения.
Сначала прочитайте одно изображение и его метаданные из .png и .csv файлов:
def read_single_disk(image_id):
""" Stores a single image to disk.
Parameters:
---------------
image_id integer unique ID for image
Returns:
----------
image image array, (32, 32, 3) to be stored
label associated meta data, int label
"""
image = np.array(Image.open(disk_dir / f"{image_id}.png"))
with open(disk_dir / f"{image_id}.csv", "r") as csvfile:
reader = csv.reader(
csvfile, delimiter=" ", quotechar="|", quoting=csv.QUOTE_MINIMAL
)
label = int(next(reader)[0])
return image, label
Reading From LMDB
Далее прочитайте тот же образ и метаданные из LMDB, открыв среду и запустив транзакцию чтения:
1 def read_single_lmdb(image_id):
2 """ Stores a single image to LMDB.
3 Parameters:
4 ---------------
5 image_id integer unique ID for image
6
7 Returns:
8 ----------
9 image image array, (32, 32, 3) to be stored
10 label associated meta data, int label
11 """
12 # Open the LMDB environment
13 env = lmdb.open(str(lmdb_dir / f"single_lmdb"), readonly=True)
14
15 # Start a new read transaction
16 with env.begin() as txn:
17 # Encode the key the same way as we stored it
18 data = txn.get(f"{image_id:08}".encode("ascii"))
19 # Remember it's a CIFAR_Image object that is loaded
20 cifar_image = pickle.loads(data)
21 # Retrieve the relevant bits
22 image = cifar_image.get_image()
23 label = cifar_image.label
24 env.close()
25
26 return image, label
Вот несколько моментов, которые следует учитывать в приведенном выше фрагменте кода:
- Строка 13: Флаг
readonly=Trueуказывает, что до завершения транзакции запись в файл LMDB не будет разрешена. На языке баз данных это эквивалентно снятию блокировки на чтение. - Строка 20: Чтобы получить объект CIFAR_Image, нужно проделать обратные шаги, которые мы предприняли для его маринования при записи. Вот здесь и пригодится
get_image()объекта.
На этом чтение изображения из LMDB завершено. Наконец, вы захотите проделать то же самое с HDF5.
Чтение из HDF5
Чтение из HDF5 очень похоже на процесс записи. Вот код для открытия и чтения файла HDF5 и разбора того же изображения и метаданных:
def read_single_hdf5(image_id):
""" Stores a single image to HDF5.
Parameters:
---------------
image_id integer unique ID for image
Returns:
----------
image image array, (32, 32, 3) to be stored
label associated meta data, int label
"""
# Open the HDF5 file
file = h5py.File(hdf5_dir / f"{image_id}.h5", "r+")
image = np.array(file["/image"]).astype("uint8")
label = int(np.array(file["/meta"]).astype("uint8"))
return image, label
Обратите внимание, что доступ к различным наборам данных в файле осуществляется путем индексирования объекта file с использованием имени набора данных, которому предшествует прямая косая черта /. Как и раньше, вы можете создать словарь, содержащий все функции чтения:
_read_single_funcs = dict(
disk=read_single_disk, lmdb=read_single_lmdb, hdf5=read_single_hdf5
)
Подготовив этот словарь, вы готовы к проведению эксперимента.
Эксперимент по чтению одного изображения
Можно было бы ожидать, что эксперимент по чтению одного изображения даст несколько тривиальные результаты, но вот код эксперимента:
from timeit import timeit
read_single_timings = dict()
for method in ("disk", "lmdb", "hdf5"):
t = timeit(
"_read_single_funcs[method](0)",
setup="image=images[0]; label=labels[0]",
number=1,
globals=globals(),
)
read_single_timings[method] = t
print(f"Method: {method}, Time usage: {t}")
Вот результаты эксперимента по чтению одного изображения:
| Method | Read Single Image + Meta |
|---|---|
| Disk | 1.61970 ms |
| LMDB | 4.52063 ms |
| HDF5 | 1.98036 ms |
Чуть быстрее считывать файлы .png и .csv непосредственно с диска, но все три метода работают тривиально быстро. Эксперименты, которые мы проведем дальше, гораздо интереснее.
Чтение многих изображений
Теперь вы можете настроить код на одновременное чтение множества изображений. Скорее всего, это действие вы будете выполнять чаще всего, поэтому производительность во время выполнения очень важна.
Настройка кода для многих изображений
Расширяя функции, описанные выше, можно создавать функции с read_many_, которые можно использовать для следующих экспериментов. Как и раньше, интересно сравнить производительность при чтении разного количества изображений, которые повторяются в коде ниже для справки:
def read_many_disk(num_images):
""" Reads image from disk.
Parameters:
---------------
num_images number of images to read
Returns:
----------
images images array, (N, 32, 32, 3) to be stored
labels associated meta data, int label (N, 1)
"""
images, labels = [], []
# Loop over all IDs and read each image in one by one
for image_id in range(num_images):
images.append(np.array(Image.open(disk_dir / f"{image_id}.png")))
with open(disk_dir / f"{num_images}.csv", "r") as csvfile:
reader = csv.reader(
csvfile, delimiter=" ", quotechar="|", quoting=csv.QUOTE_MINIMAL
)
for row in reader:
labels.append(int(row[0]))
return images, labels
def read_many_lmdb(num_images):
""" Reads image from LMDB.
Parameters:
---------------
num_images number of images to read
Returns:
----------
images images array, (N, 32, 32, 3) to be stored
labels associated meta data, int label (N, 1)
"""
images, labels = [], []
env = lmdb.open(str(lmdb_dir / f"{num_images}_lmdb"), readonly=True)
# Start a new read transaction
with env.begin() as txn:
# Read all images in one single transaction, with one lock
# We could split this up into multiple transactions if needed
for image_id in range(num_images):
data = txn.get(f"{image_id:08}".encode("ascii"))
# Remember that it's a CIFAR_Image object
# that is stored as the value
cifar_image = pickle.loads(data)
# Retrieve the relevant bits
images.append(cifar_image.get_image())
labels.append(cifar_image.label)
env.close()
return images, labels
def read_many_hdf5(num_images):
""" Reads image from HDF5.
Parameters:
---------------
num_images number of images to read
Returns:
----------
images images array, (N, 32, 32, 3) to be stored
labels associated meta data, int label (N, 1)
"""
images, labels = [], []
# Open the HDF5 file
file = h5py.File(hdf5_dir / f"{num_images}_many.h5", "r+")
images = np.array(file["/images"]).astype("uint8")
labels = np.array(file["/meta"]).astype("uint8")
return images, labels
_read_many_funcs = dict(
disk=read_many_disk, lmdb=read_many_lmdb, hdf5=read_many_hdf5
)
Если функции чтения хранятся в словаре, как и функции записи, то все готово к эксперименту.
Эксперимент по чтению большого количества изображений
Теперь вы можете провести эксперимент по считыванию большого количества изображений:
from timeit import timeit
read_many_timings = {"disk": [], "lmdb": [], "hdf5": []}
for cutoff in cutoffs:
for method in ("disk", "lmdb", "hdf5"):
t = timeit(
"_read_many_funcs[method](num_images)",
setup="num_images=cutoff",
number=1,
globals=globals(),
)
read_many_timings[method].append(t)
# Print out the method, cutoff, and elapsed time
print(f"Method: {method}, No. images: {cutoff}, Time usage: {t}")
Как мы уже делали ранее, вы можете построить график результатов эксперимента по чтению:
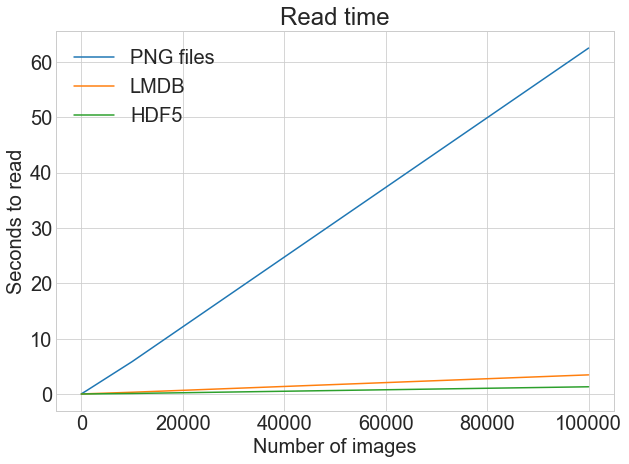
На верхнем графике показано обычное, не скорректированное время чтения, демонстрирующее резкую разницу между чтением из файлов .png и LMDB или HDF5.
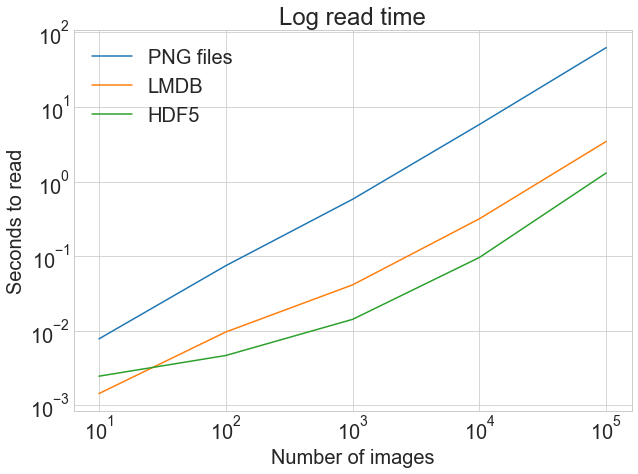
Напротив, график внизу показывает log тайминги, подчеркивая относительную разницу при меньшем количестве изображений. А именно, мы видим, как HDF5 начинает отставать, но с увеличением количества изображений становится стабильно быстрее LMDB с небольшим отрывом.
Используя ту же функцию построения, что и для таймингов записи, мы получили следующее:
disk_x_r = read_many_timings["disk"]
lmdb_x_r = read_many_timings["lmdb"]
hdf5_x_r = read_many_timings["hdf5"]
plot_with_legend(
cutoffs,
[disk_x_r, lmdb_x_r, hdf5_x_r],
["PNG files", "LMDB", "HDF5"],
"Number of images",
"Seconds to read",
"Read time",
log=False,
)
plot_with_legend(
cutoffs,
[disk_x_r, lmdb_x_r, hdf5_x_r],
["PNG files", "LMDB", "HDF5"],
"Number of images",
"Seconds to read",
"Log read time",
log=True,
)
На практике время записи часто менее критично, чем время чтения. Представьте, что вы обучаете глубокую нейронную сеть на изображениях, а в оперативную память помещается только половина всего набора данных изображений за один раз. Для каждой эпохи обучения сети требуется весь набор данных, а для сходимости модели необходимо несколько сотен эпох. По сути, вы будете считывать половину набора данных в память каждую эпоху.
Есть несколько трюков, которые люди делают, например, тренируют псевдоэпохи, чтобы сделать это немного лучше, но вы поняли идею.
Теперь снова посмотрите на график чтения выше. Разница между 40-секундным и 4-секундным временем чтения - это разница между шестью часами ожидания обучения модели и сорока минутами!
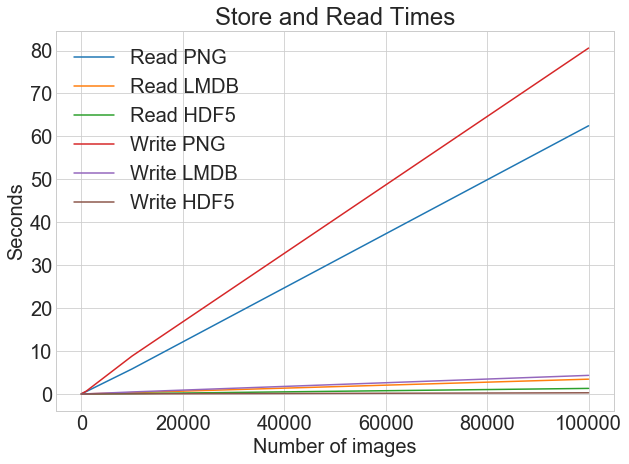
Если мы рассмотрим время чтения и записи на одном графике, то получим следующее:
Вы можете построить все тайминги чтения и записи на одном графике, используя одну и ту же функцию построения:
plot_with_legend(
cutoffs,
[disk_x_r, lmdb_x_r, hdf5_x_r, disk_x, lmdb_x, hdf5_x],
[
"Read PNG",
"Read LMDB",
"Read HDF5",
"Write PNG",
"Write LMDB",
"Write HDF5",
],
"Number of images",
"Seconds",
"Log Store and Read Times",
log=False,
)
При хранении изображений в виде файлов .png существует большая разница между временем записи и чтения. Однако при использовании LMDB и HDF5 разница гораздо менее заметна. В целом, даже если время чтения более критично, чем время записи, есть весомые аргументы в пользу хранения изображений с помощью LMDB или HDF5.
Теперь, когда вы убедились в преимуществах производительности LMDB и HDF5, давайте посмотрим на другой важный показатель: использование диска.
Рассмотрение использования диска
Скорость - не единственная метрика производительности, которая может вас интересовать. Мы уже имеем дело с очень большими массивами данных, поэтому дисковое пространство также является весьма обоснованной и актуальной проблемой.
Предположим, у вас есть набор данных изображений объемом 3 ТБ. Предположительно, в отличие от нашего примера с CIFAR, они уже находятся где-то на диске, поэтому, используя альтернативный метод хранения, вы, по сути, создаете их копию, которую также необходимо сохранить. Это даст вам огромный выигрыш в производительности при использовании образов, но вам нужно убедиться, что у вас достаточно места на диске.
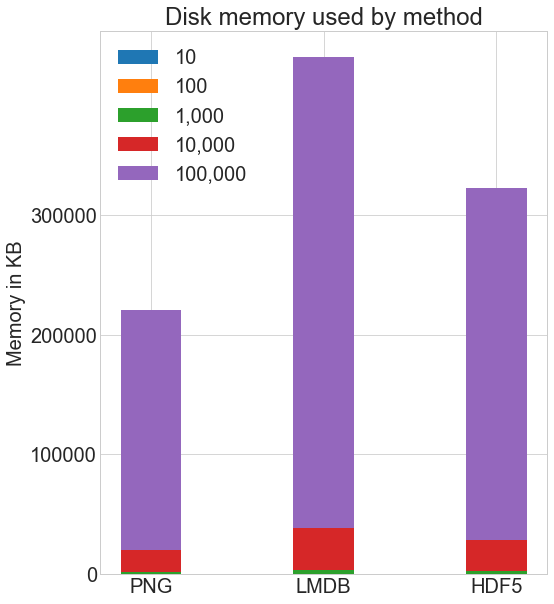
Сколько дискового пространства используют различные методы хранения? Вот дисковое пространство, используемое каждым методом для каждого количества изображений:
Я использовал команду Linux du -h -c folder_name/*, чтобы вычислить использование диска в моей системе. В этом методе присутствует некоторое приближение из-за округления, но вот общее сравнение:
# Memory used in KB
disk_mem = [24, 204, 2004, 20032, 200296]
lmdb_mem = [60, 420, 4000, 39000, 393000]
hdf5_mem = [36, 304, 2900, 29000, 293000]
X = [disk_mem, lmdb_mem, hdf5_mem]
ind = np.arange(3)
width = 0.35
plt.subplots(figsize=(8, 10))
plots = [plt.bar(ind, [row[0] for row in X], width)]
for i in range(1, len(cutoffs)):
plots.append(
plt.bar(
ind, [row[i] for row in X], width, bottom=[row[i - 1] for row in X]
)
)
plt.ylabel("Memory in KB")
plt.title("Disk memory used by method")
plt.xticks(ind, ("PNG", "LMDB", "HDF5"))
plt.yticks(np.arange(0, 400000, 100000))
plt.legend(
[plot[0] for plot in plots], ("10", "100", "1,000", "10,000", "100,000")
)
plt.show()
Как HDF5, так и LMDB занимают больше места на диске, чем при хранении обычных .png изображений. Важно отметить, что использование диска и производительность LMDB и HDF5 сильно зависят от различных факторов, включая операционную систему и, что более важно, размер данных, которые вы храните.
LMDB достигает своей эффективности за счет кэширования и использования преимуществ размеров страниц ОС. Вам не нужно разбираться в его внутреннем устройстве, но учтите, что при больших изображениях вы будете значительно больше использовать диск с LMDB, потому что изображения не поместятся на листовых страницах LMDB, обычном месте хранения в дереве, и вместо этого у вас будет много переполненных страниц. Полоса LMDB на приведенном выше графике будет уходить за пределы графика.
Наши изображения размером 32x32x3 пикселя относительно малы по сравнению со средними изображениями, которые вы можете использовать, и они обеспечивают оптимальную производительность LMDB.
Хотя мы не будем исследовать это экспериментально, по моему собственному опыту с изображениями 256x256x3 или 512x512x3 пикселей HDF5 обычно немного эффективнее с точки зрения использования диска, чем LMDB. Это хороший переход к заключительному разделу, качественному обсуждению различий между методами.
Дискуссия
Есть и другие отличительные особенности LMDB и HDF5, о которых стоит знать, а также важно кратко обсудить некоторые критические замечания в адрес обоих методов. Если вы захотите узнать больше, вместе с обсуждением вы найдете несколько ссылок.
Параллельный доступ
Ключевое сравнение, которое мы не проверяли в экспериментах выше, - это одновременное чтение и запись. Часто при работе с такими большими массивами данных может потребоваться ускорение работы за счет распараллеливания.
В большинстве случаев вы не будете заинтересованы в одновременном чтении частей одного и того же изображения, но вы захотите читать несколько изображений одновременно. При таком определении параллелизма хранение на диске в виде .png файлов фактически обеспечивает полный параллелизм. Ничто не мешает вам читать несколько изображений одновременно из разных потоков или записывать несколько файлов одновременно, если только имена изображений различны.
Как насчет LMDB? В среде LMDB одновременно может быть несколько читателей, но только один писатель, и писатели не блокируют читателей. Подробнее об этом можно прочитать на сайте технологии LMDB.
Множество приложений могут одновременно обращаться к одной и той же базе данных LMDB, а несколько потоков одного процесса также могут одновременно обращаться к LMDB для чтения. Это позволяет еще больше ускорить процесс чтения: если разделить весь CIFAR на десять наборов, то можно настроить десять процессов на чтение каждого набора, и это разделит время загрузки на десять.
HDF5 также предлагает параллельный ввод/вывод, позволяя одновременное чтение и запись. Однако при реализации удерживается блокировка записи, и доступ осуществляется последовательно, если у вас нет параллельной файловой системы.
Если вы работаете в такой системе, есть два основных варианта, которые более подробно рассматриваются в этой статье HDF Group о параллельном вводе-выводе. Все может оказаться довольно сложным, и самый простой вариант - грамотно разбить набор данных на несколько файлов HDF5, чтобы каждый процесс мог работать с одним .h5 файлом независимо от других.
Документация
Если вы набираете в Google lmdb, то, по крайней мере в Великобритании, третьим результатом поиска будет IMDb, база данных фильмов в Интернете. Это не то, что вы искали!
На самом деле, существует один основной источник документации для Python-вязки LMDB, который размещен на сайте Read the Docs LMDB. Хотя пакет Python еще не достиг версии > 0.94, он достаточно широко используется и считается стабильным.
Что касается самой технологии LMDB, то более подробная документация находится на веб-сайте технологии LMDB, который может показаться немного похожим на изучение калькуляции во втором классе, если только вы не начнете с их страницы Getting Started.
Для HDF5 есть очень понятная документация на сайте h5py docs, а также полезная запись в блоге Кристофера Ловелла, которая представляет собой отличный обзор того, как использовать пакет h5py. Книга O'Reilly "Python и HDF5" также является хорошим способом начать работу.
Хотя и LMDB, и HDF5 не так документированы, как хотелось бы новичку, у них большие сообщества пользователей, поэтому более глубокий поиск в Google обычно дает полезные результаты.
Более критический взгляд на реализацию
В системах хранения данных нет утопии, и как LMDB, так и HDF5 имеют свою долю подводных камней.
Ключевым моментом в работе LMDB является то, что новые данные записываются без перезаписи или перемещения существующих данных. Это конструктивное решение, которое позволяет добиться чрезвычайно быстрого чтения, что вы наблюдали в наших экспериментах, а также гарантирует целостность и надежность данных без дополнительной необходимости ведения журналов транзакций.
Помните, однако, что вам нужно было определить map_size параметр для выделения памяти перед записью в новую базу данных? Именно здесь LMDB может доставить немало хлопот. Предположим, вы создали базу данных LMDB, и все замечательно. Вы терпеливо ждали, пока ваш огромный набор данных будет упакован в LMDB.
Потом, позже, вы вспомните, что вам нужно добавить новые данные. Даже с буфером, который вы указали в map_size, вы вполне можете ожидать появления ошибки lmdb.MapFullError. Если вы не хотите переписать всю свою базу данных с обновленными map_size, вам придется хранить эти новые данные в отдельном файле LMDB. Даже если одна транзакция может охватывать несколько LMDB-файлов, наличие нескольких файлов все равно может стать проблемой.
Кроме того, некоторые системы имеют ограничения на объем памяти, который может быть востребован одновременно. По моему собственному опыту, при работе с высокопроизводительными вычислительными системами (HPC) это оказалось крайне неприятным и часто заставляло меня предпочесть HDF5 вместо LMDB.
Как в LMDB, так и в HDF5, в память сразу считывается только запрашиваемый элемент. В LMDB пары ключ-единица считываются в память по одному, а в HDF5 к объекту dataset можно обращаться как к массиву Python, с индексацией dataset[i], диапазонами, dataset[i:j] и прочим сращиванием dataset[i:j:interval].
Из-за того, что системы оптимизированы, и в зависимости от вашей операционной системы, порядок доступа к элементам может влиять на производительность.
По моему опыту, в целом верно, что для LMDB можно получить лучшую производительность при последовательном доступе к элементам по ключу (пары ключ-значение хранятся в памяти в алфавитно-цифровом порядке по ключу), а для HDF5 доступ к большим диапазонам будет лучше, чем чтение каждого элемента набора данных по одному с помощью следующего:
# Slightly slower
for i in range(len(dataset)):
# Read the ith value in the dataset, one at a time
do_something_with(dataset[i])
# This is better
data = dataset[:]
for d in data:
do_something_with(d)
Если вы раздумываете над выбором формата хранения файлов для написания программного обеспечения, было бы упущением не упомянуть Moving away from HDF5 от Cyrille Rossant о подводных камнях HDF5, и ответ Konrad Hinsen On HDF5 and the future of data management, который показывает, как можно избежать некоторых подводных камней в его собственных случаях использования с множеством небольших наборов данных, а не несколькими огромными. Обратите внимание, что относительно небольшой набор данных все равно имеет размер в несколько ГБ.
Интеграция с другими библиотеками
Если вы имеете дело с действительно большими наборами данных, весьма вероятно, что вы будете делать с ними что-то значительное. Стоит подумать о библиотеках глубокого обучения и об интеграции с LMDB и HDF5.
Прежде всего, все библиотеки поддерживают чтение изображений с диска в виде файлов .png при условии, что вы преобразуете их в массивы NumPy ожидаемого формата. Это справедливо для всех методов, и мы уже видели выше, что считывать изображения в виде массивов относительно просто.
Вот несколько наиболее популярных библиотек глубокого обучения и их интеграция с LMDB и HDF5:
-
Caffe имеет стабильную, хорошо поддерживаемую интеграцию LMDB, и она обрабатывает шаг чтения прозрачно. Слой LMDB также может быть легко заменен базой данных HDF5.
-
Keras использует формат HDF5 для сохранения и восстановления моделей. Из этого следует, что TensorFlow также может это делать.
-
TensorFlow имеет встроенный класс
LMDBDataset, который предоставляет интерфейс для чтения входных данных из файла LMDB и может производить итераторы и тензоры в пакетном режиме. TensorFlow не имеет встроенного класса для HDF5, но его можно написать, наследуя от классаDataset. Лично я использую собственный класс, созданный для оптимального доступа к чтению на основе того, как я структурирую свои HDF5-файлы. -
Theano не поддерживает какой-либо конкретный формат файлов или баз данных, но, как уже говорилось ранее, может использовать все, что угодно, если только оно считывается как N-мерный массив.
Хотя это далеко не полный обзор, мы надеемся, что он даст вам представление об интеграции LMDB/HDF5 с некоторыми ключевыми библиотеками глубокого обучения.
Несколько личных соображений о хранении изображений на Python
В своей повседневной работе, анализируя терабайты медицинских изображений, я использую как LMDB, так и HDF5, и понял, что при любом способе хранения данных крайне важна продуманность.
Часто модели необходимо обучать с помощью k-fold cross validation, что предполагает разбиение всего набора данных на k наборов (k обычно равно 10), и обучение k моделей, каждая из которых используется в качестве тестового набора на разных k наборах. Это гарантирует, что модель не будет чрезмерно соответствовать набору данных или, другими словами, не сможет делать хорошие предсказания на невидимых данных.
Стандартный способ создания k-набора заключается в том, чтобы поместить в каждый k-набор равное количество данных каждого типа, представленных в наборе данных. Таким образом, сохранение каждого k-набора в отдельном наборе данных HDF5 повышает эффективность. Иногда один k-набор не может быть загружен в память сразу, поэтому даже упорядочивание данных в наборе данных требует некоторой предусмотрительности.
В случае с LMDB я также стараюсь заранее планировать создание базы данных. Перед сохранением изображений стоит задать несколько хороших вопросов:
- Как сохранить изображения таким образом, чтобы большинство чтений были последовательными?
- Какие бывают хорошие ключи?
- Как рассчитать хороший
map_size, предвидя возможные будущие изменения в наборе данных? - Насколько большой может быть одна транзакция, и как следует делить транзакции на части?
Независимо от способа хранения, когда вы имеете дело с большими наборами данных изображений, немного планирования не помешает.
Заключение
Вы добрались до конца! Теперь вы можете взглянуть на большую тему с высоты птичьего полета.
В этой статье вы познакомились с тремя способами хранения и доступа к множеству изображений в Python и, возможно, имели возможность поиграть с некоторыми из них. Весь код для этой статьи находится в Jupyter notebook здесь или Python script здесь. Запускайте на свой страх и риск, поскольку несколько гигабайт вашего дискового пространства будут заняты маленькими квадратными изображениями автомобилей, лодок и так далее.
Вы уже видели, как различные методы хранения могут радикально влиять на время чтения и записи, а также некоторые плюсы и минусы трех методов, рассмотренных в этой статье. Хотя хранение изображений в виде файлов .png может быть наиболее интуитивно понятным, есть большие преимущества в производительности, если рассматривать такие методы, как HDF5 или LMDB.
Не стесняйтесь обсуждать в разделе комментариев отличные методы хранения данных, не рассмотренные в этой статье, такие как LevelDB, Feather, TileDB, Badger, BoltDB, или любые другие. Не существует идеального метода хранения данных, и выбор оптимального метода зависит от конкретного набора данных и условий использования.
Дальнейшее чтение
Вот некоторые ссылки, относящиеся к трем методам, рассмотренным в этой статье:
- Связывание Python для LMDB
- Документация по LMDB: Начало работы
- Привязка Python для HDF5 (h5py)
- The HDF5 Group
- "Python и HDF5" от O'Reilly
- Подушка
Вам также может понравиться "Анализ систем хранения изображений для масштабируемого обучения глубоких нейронных сетей" Лима, Янга и Паттона. В этой статье рассматриваются эксперименты, аналогичные тем, что описаны в данной статье, но в гораздо большем масштабе, с учетом холодного и теплого кэша, а также других факторов.
Back to Top